我的数据
“我的数据”可查看账号下可用于创建分析流程的数据,包括寻因关联标准分析数据及用户自行上传的数据。
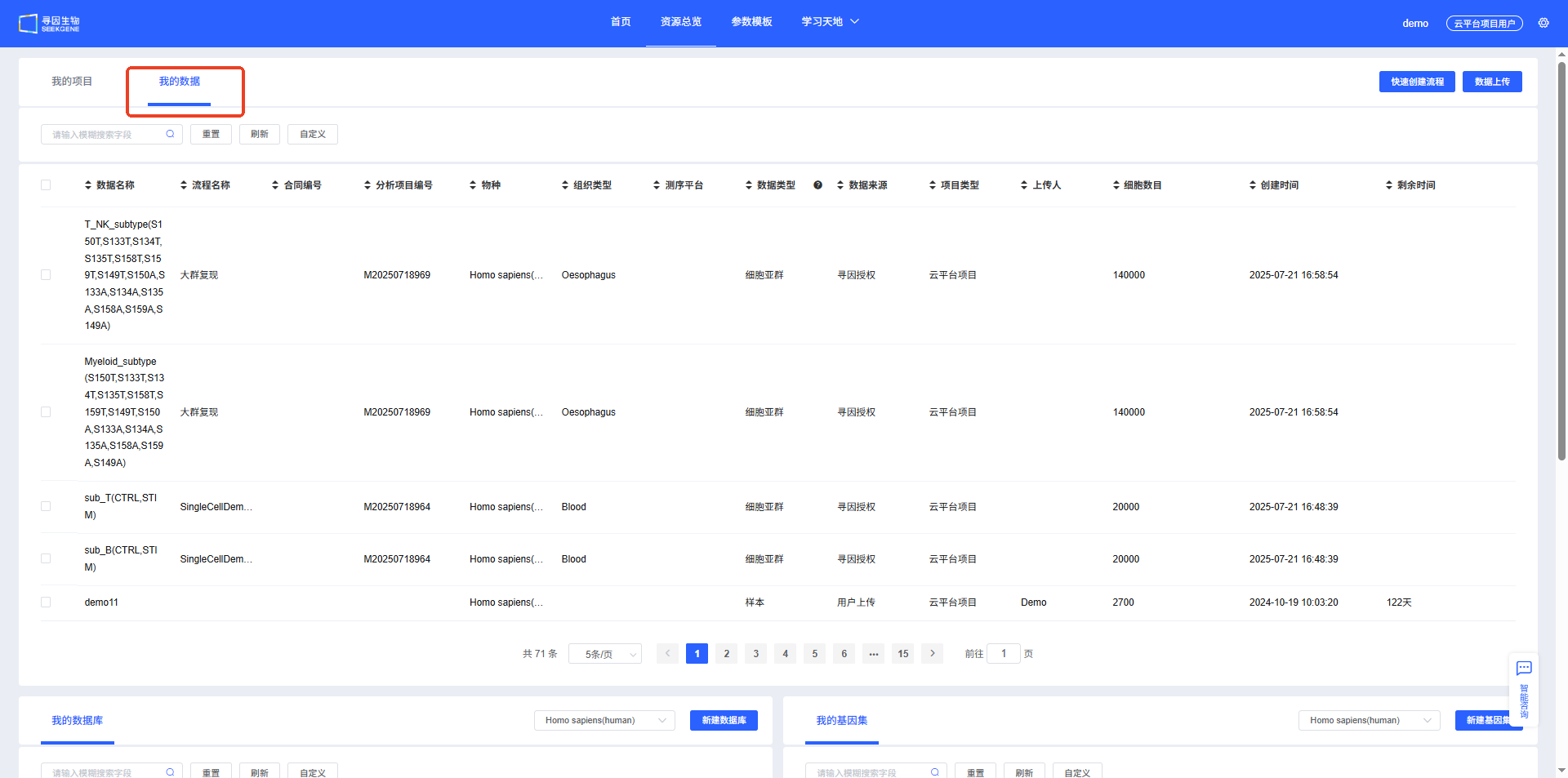
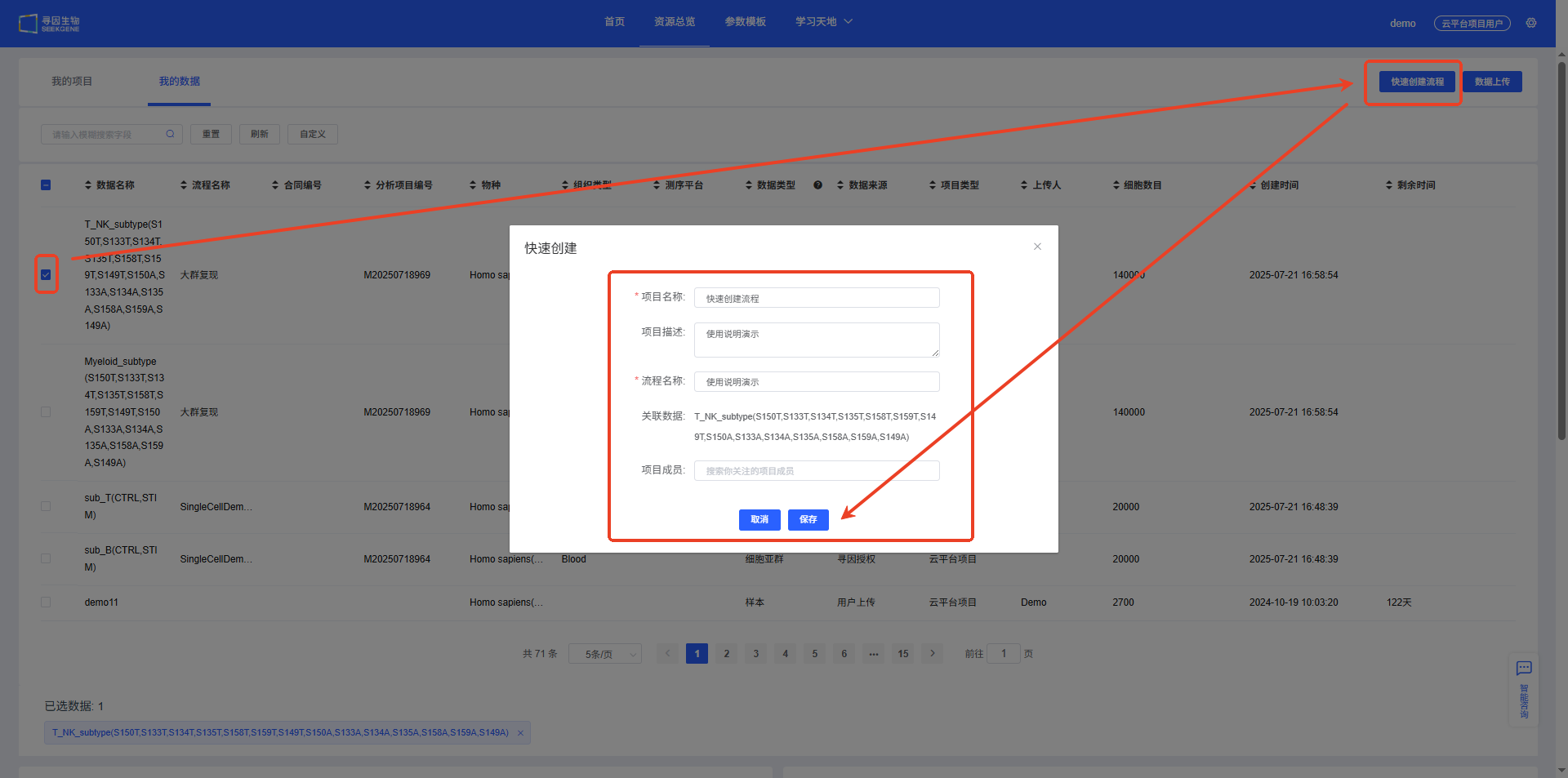
快速创建流程
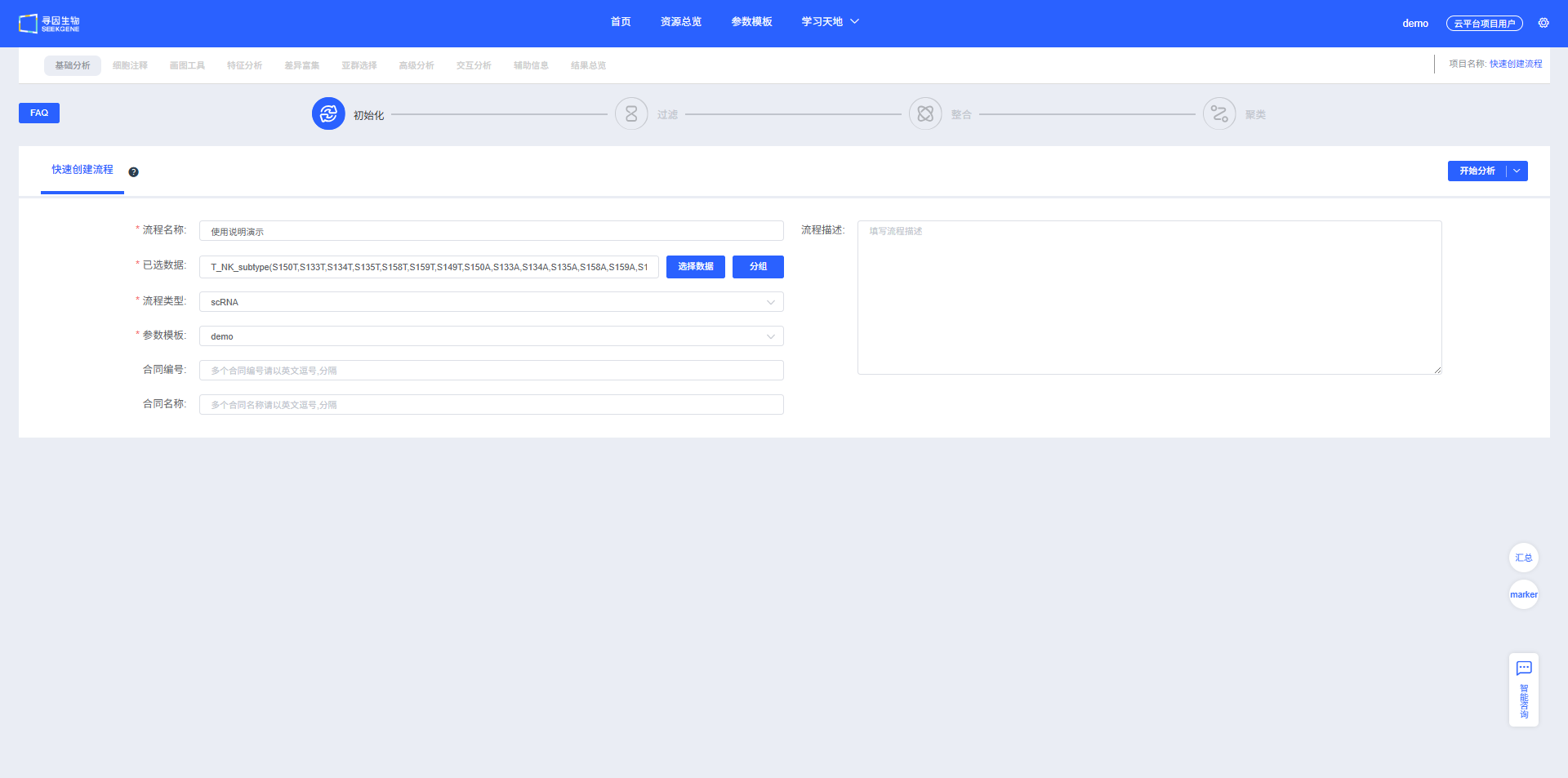
选择需要分析的样本数据,点击【快速创建流程】按钮可快速创建项目及其分析流程,保存后会跳转到新项目流程进行初始化,后续步骤可参考前文“基础分析”。
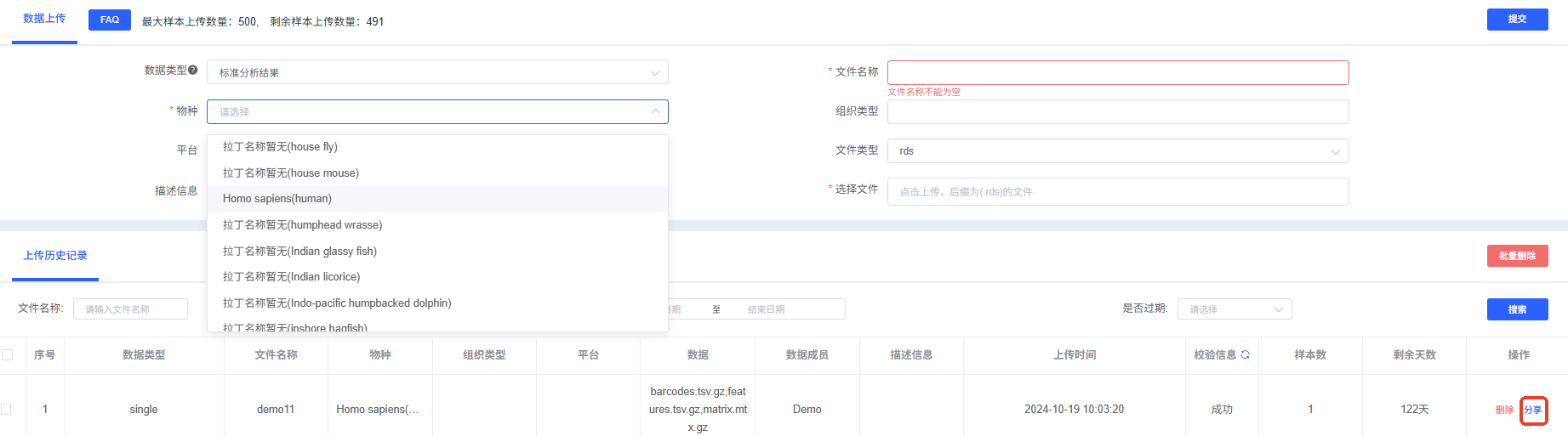
数据上传
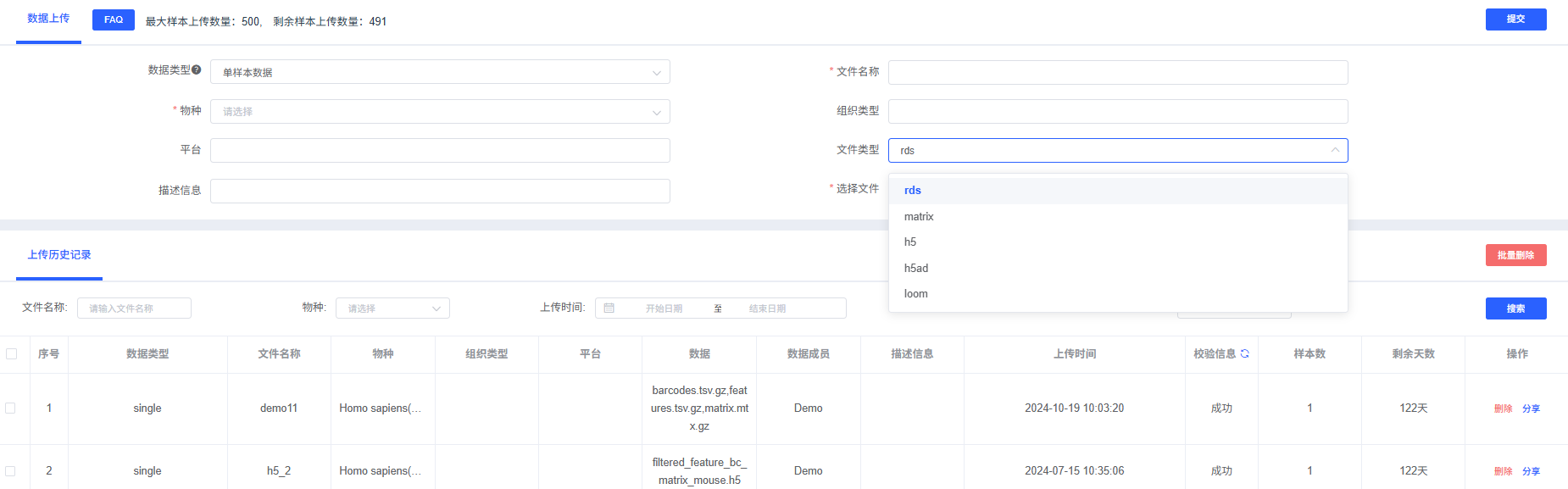
数据上传主要用于本地上传需分析的单细胞数据,点击该按钮会跳转到数据上传窗口,根据信息填写选择数据上传,数据类型分为标准分析和单样本数据两种。
标准分析结果
标准分析结果指已基于 Seurat 或 scanpy 完成单细胞标准分析(过滤、标准化、归一化、降维(整合)、聚类等分析)的文件,仅支持 SeuratObject (rds) 格式数据。
1.1 上传“标准分析结果”的“rds”类型文件,填写信息并点击【提交】,待校验信息显示“成功”后可创建流程进行分析,还可将数据“分享”给其他用户。
NOTE
上传标准分析结果可在基础分析阶段跳过过滤整合。
1.2 数据类型为“标准分析结果”的“rds”类型文件格式要求:
- 数据结构必须为
SeuratObject(rds) 对象格式,文件必须以.rds结尾,metadata 中必须包括nCount_RNA、nFeature_RNA和RNA_snn_res.x.x(或resolution.x.x,x.x 为分辨率值,如RNA_snn_res.0.5或resolution.0.5)标签。 - 若 metadata 中无
Sample标签,则以上传时的文件名称作为样本名称。 - 若 metadata 中无
percent.mito信息,将自动计算以MT-、mt-或Mt-为起始的基因名占全部基因的比例,并以mito为列名。 - 检验信息为“成功”时,说明上传数据成功。
| nCount_RNA(必有) | nFeature_RNA(必有) | RNA_snn_res.0.5(必有) | Sample(多样本时必有) | percent.mt(可选) | |
|---|---|---|---|---|---|
| AAACATACAACCAC-1 | 2419 | 779 | 4 | A | 3.0177759 |
| AAACATACAACTAC-1 | 4219 | 1727 | 3 | A | 0.8897363 |
| AAACATACAACGAC-1 | 980 | 545 | 2 | C | 1.2244898 |
| AAACATACAACCCC-1 | 263 | 132 | 4 | C | 1.6643551 |
1.3 云平台如何读取“标准分析结果”的“rds”类型文件:
# 加载 R 包
library(Seurat)
# 读取数据
standard_demo_rds <- readRDS("standard_demo.rds")1.4 文件核验未通过的常见原因:
- 标准分析结果 rds 格式文件并非
SeuratObject(rds) 类型。 - metadata 中未检测到
nCount_RNA、nFeature_RNA信息。 - metadata 中无
RNA_snn_res.x.x或resolution.x.x分辨率标签。 - metadata 中无
Sample标签,导致多样本数据被视为单样本。
单样本数据
单样本数据指单细胞的表达矩阵文件,支持 SeuratObject (rds)、数据矩阵(matrix)、HDF5 (h5)、Scanpy 文件(h5ad)和基于 HDF5 的 loom 文件。
2.1 上传“单样本数据”可支持多种格式,包括“rds”、“matrix”、“h5”、“h5ad”和“loom”类型文件,填写信息并点击【提交】,待校验信息显示“成功”后可创建流程进行分析,还可将数据“分享”给其他用户。
2.2 云平台如何读取“单样本数据”的“rds”类型文件:
# 加载 R 包
library(Seurat)
# 读取数据
single_demo_rds <- readRDS("single_demo.rds")2.3 “matrix”类型文件格式要求:
- 包含
barcodes.tsv(1 列 barcode)、features.tsv(2 列,ensemble id 和基因名)、matrix.mtx(表头和 3 列,分别为基因名、barcode、counts)。
barcodes.tsv 示例:
AAACATACAACCAC-1
AAACATTGAGCTAC-1
...features.tsv 示例:
ENSG00000243485 MIR1302-10
ENSG00000237613 FAM138A
...matrix.mtx 示例:
%%MatrixMarket matrix coordinate real general
%
32738 2700 2286884
32709 1 4
...2.4 云平台如何读取“matrix”类型文件:
# 加载 R 包
library(Seurat)
# 读取数据
dir = "./single_demo_matirx"
single_demo_matrix = CreateSeuratObject(Read10X(dir))2.5 云平台如何读取“h5”类型文件:
# 加载 R 包
library(Seurat)
# 读取数据
single_demo_h5 <- CreateSeuratObject(Read10X_h5("single_demo.h5"))2.6 云平台如何读取“h5ad”类型文件:
# 加载 R 包
library(SeuratDisk)
# 读取数据
Convert('single_demo.h5ad', "h5seurat", overwrite = TRUE, assay = "RNA")
single_demo_h5seurat <- LoadH5Seurat("single_demo.h5seurat")CAUTION
h5ad 数据列应为分类变量,否则执行 LoadH5Seurat 时会报错 Missing required datasets 'levels' and 'values' 导致上传失败。
2.7 云平台如何读取“loom”类型文件:
# 加载 R 包
library(SeuratDisk)
# 读取数据
single_demo_loom <- LoadLoom("single_demo.loom")